الڤيروس التاجي المرتبط بالمتلازمة التنفسية الحادة الوخيمة
| الڤيروس التاجي المرتبط بالمتلازمة التنفسية الحادة الوخيمة | |
|---|---|
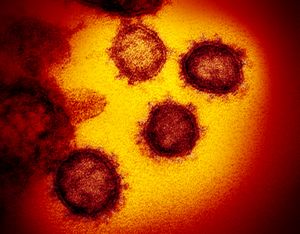
| |
| Transmission electron micrograph of SARS-related coronaviruses emerging from host cells cultured in the lab | |
| تصنيف الفيروسات | |
| نطاق غير مصنف: | الڤيروسات |
| المملكة: | ريبوڤيريا |
| تحت جنس: | Sarbecovirus |
| Species: | الڤيروس التاجي المرتبط بالمتلازمة التنفسية الحادة الوخيمة
|
| سلالات | |
|
SARS-CoV
| |
| Synonyms | |
| |
الفيروس التاجي الحاد المرتبط بمتلازمة الجهاز التنفسي الحادة ( SARSr-CoV ))[note 1] و أحد أنواع الفيروسات التاجية التي تصيب البشر والخفافيش وبعض الثدييات الأخرى.[2][3] وهو فيروس حمض ريبي نووي مفرد السلسلة موجب الاتجاه مغلف الذي يدخل الخلية المضيفة عن طريق الارتباط بمستقبل إلانزيم محول للأنجيوتنسين 2[4] و هو عضو في فيروس كورونا بيتا والجنس الفرعي فيروس سارب كورونا .[5][6]
تسببت سلالتين من الفيروس لحالة تفشي شديدة لأمراض الجهاز التنفسي في الإنسان: فيروس كورونا المرتبط بالمتلازمة التنفسية الحادة الوخيمة ، وهو ما تسبب في تفشي لمتلازمة تنفسية حادة وخيمة (سارس) بين عامي 2002 و 2003، و السارس COV-2 (فيروس كورونا 2 المرتبط بالمتلازمة التنفسية الحادة الشديدة) الذي منذ أواخر عام 2019 وتسبب جائحة مرض فيروس كورونا 2019 (COVID-19).[7] هناك المئات من سلالات أخرى من (الفيروس التاجي الحاد المرتبط بمتلازمة الجهاز التنفسي الحادة) SARSr-CoV ، وكلها معروف فقط لإصابة الأنواع غير البشرية: الخفافيش هي المستودع الرئيسي للعديد من سلالات الفيروسات التاجية المرتبطة بالسارس ، وتم التعرف على العديد من السلالات في الزباد ، والتي كانت أسلاف المحتمل من مرض السارس - CoV.[7][8]
ان الفيروس التاجي المرتبط بالسارس واحداً من عدة فيروسات حددتها منظمة الصحة العالمية في عام 2016 كسبب محتمل لوباء مستقبلي في خطة جديدة وضعت بعد وباء إيبولا للبحث والتطوير العاجل قبل وأثناء الوباء نحو تشخيص الاختبارات واللقاحات والأدوية. جاء التوقع مع جائحة ڤيروس كورونا 2019-2020.[9][10]
التصنيف
الڤيروس التاجي المرتبط بالمتلازمة التنفسية الحادة الوخيمة هو عضو في جنس فيروس كورونا بيتا (المجموعة 2) والجنس الفرعي فيروس سارب كورونا (المجموعة الفرعية B).[11] فيروس سارب كورونا ، على عكس ايمبيكو فيروس أو فيروس كورونا ألفا ، لديها بروتيناز واحد شبيه بالبابين فقط (PLpro) بدلاً من اثنين في إطار القراءة المفتوح ORF1.[12] تم تحديد الفيروس التاجي الحاد المرتبط بمتلازمة الجهاز التنفسي الحادة على أنه انفصال مبكر عن فيروسات بيتاكورونا بناءً على مجموعة من المجالات المحفوظة التي تشاركها مع المجموعة..[13][14]
تعمل الخفافيش كمستضيف رئيسي للفيروس التاجي الحاد المرتبط بالمتلازمة الجهاز التنفسي الحادة. وقد توغل الفيروس في المستضيف الرئيسي (الخفافيش) لفترة طويلة من الزمن.[15] في الآونة الأخيرة فقط تطورت سلالات من الڤيروس التاجي للمتلازمة التنفسية الحادة الوخيمة(SARS-CoV) وجعل الانتقال عبر الأنواع تقفز من الخفافيش إلى البشر ، كما هو الحال في سلالات الڤيروس التاجي للمتلازمة التنفسية الحادة الوخيمة و ڤيروس كورونا 2 المرتبط بالمتلازمة التنفسية الحادة الوخيمة(SARS-CoV-2).[16][17] تنحدر هذه السلالات من سلف واحد ولكنها جعلت الانتقال عبر الانواع يقفز إلى البشر بشكل منفصل. ليس ڤيروس كورونا 2 المرتبط بمتلازمة التنفسية الحادة الوخيمة (SARS-CoV-2) سليلًا مباشرًا للفيروس التاجي الحاد المرتبط بمتلازمة الجهاز التنفسي الحادة(SARS-CoV)..[7]
الجينوم
إن الفيروس التاجي الحاد المرتبط بمتلازمة الجهاز التنفسي الحادة هو فيروس رنا ، مفرد السلسلة موجب الاتجاه مغلف . الجينوم الخاص به هو حوالي 30 زوج قاعدي ، وهو واحد من أكبر فيروسات الحمض الريبي النووي . يحتوي الفيروس على 14 قالب قراءة مفتوح يتداخل في بعض الحالات.[18] يحتوي الجينوم على غطاء 5′ميثيل و 3 ′ ذيل عديد الادنينلات.[19] يوجد 265 نيوكليوتايدات في منطقة 5' غير مترجمة و 342 نيوكليوتيدات في منطقة 3' غير مترجمة.[18]
يسمح الغطاء 5' ميثيل و 3 'الذيل متعدد الأدينيلات مرسال الحمض النووي الريبي بترجمة مباشرة من الريبوسوم في الخلية المضيفة عند دخول الفيروس.[20] يتشابه SARSr-CoV (الفيروس التاجي الحاد المرتبط بالمتلازمة الجهاز التنفسي الحادة) مع الفيروسات التاجية الأخرى في أن تعبير الجينوم يبدأ بالترجمة بواسطة الريبوسومات في الخلية المضيفة لقالبي القراءة المفتوحين المتداخلين (ORFs) الأوليين ، 1 أ و 1 ب ، وكلاهما ينتج بروتينات متعددة.[18]
| وظيفة الڤيروس التاجي للمتلازمة التنفسية الحادة الوخيمة بروتينات الجينوم (orf1a to orf9b) | |
|---|---|
| البروتين | الوظيفة[21][22][23] |
| orf1a, orf1b |
النسخ/البروتينات المنتسخة
(pp1ab) |
| orf2 | الشوكة (S) بروتين, التصاق الفيروس ودخوله (بروتينات هيكلية) |
| orf3a | تتفاعل مع S, E, M بروتينات هيكلية; نشاط قناة الأيون; ينظم السيتوكينات والكيموكينات مثل IL-8 و RANTES; ينظم NF-κB و JNK; تحفز الاستموات و توقف دورة الخلية, خلال Caspase 8 و -9, وبواسطة Bax, p53, و p38 MAP kinase |
| orf3b | ينظم السيتوكينات والكيميوكينات بواسطة RUNX1b; يمنع إنتاج إنترفيرون من النمط الأول IFN; يحفز الاستموات وتوقف دورة الخلية |
| orf4 | الغلاف (E) البروتين وتجميع الفيروسات والتبرعم
(بروتين هيكلي) |
| orf5 | غشاء (M) البروتين ، وتجميع الفيروسات والتبرعم
(بروتين هيكلي) |
| orf6 | يعزز تصنيع الدنا (DNA).
; |
| orf7a | يثبط تخليق البروتين الخلوي; يحفز الاستجابة الالتهابية عن طريق NF-kappaB و انترلوكين -8 المعزز; ينظم كيموكينات مثل انترلوكين-8 و RANTES; ينظم JNK, p38 MAP كيناز; يحفز الاستموات وتوقف دورة الخلية |
| orf7b | غير معروف |
| orf8a | يحفز الاستموات عن طريق مسار المتقدرة |
| orf8b | يعزز تصنيع الحمض النووي الخلوي (DNA) |
| orf9a | قفيصة منواة (N) بروتين ، تغليف رنا (RNA) الفيروسي |
| orf9b | تحفز الاستموات |
وظائف العديد من البروتينات الفيروسية معروفة.[24] ترمز ORFs 1a و 1 b إلى بروتين متعدد النسخ المتماثل / الاستنساخ ، وفي وقت لاحق يرمز ORFs 2 و 4 و 5 و 9a ، على التوالي ، البروتينات الهيكلية الرئيسية الأربعة: الشوكة ، الغلاف ، الغشاء ، و قفيصة منواة.[25] ترميز ORFs اللاحقة أيضًا لثمانية بروتينات فريدة (orf3a إلى orf9b) ، والمعروفة باسم البروتينات الإضافية ، والكثير منها بدون تجانسات معروفة. الوظائف المختلفة للبروتينات الإضافية ليست مفهومة جيدًا.[24]
الشكل
إن لشكل الفيروس التاجي المرتبط بالسارس(الفيروس التاجي الحاد المرتبط بمتلازمة الجهاز التنفسي الحادة) سمة مميزة لعائلة الفيروس التاجي ككل. والفيروسات عبارة عن جزيئات كروية كبيرة متعددة الأشكال ذات إسقاطات بصليّة منتفخة تشكل هالة حول الجسيمات في المجاهر الإلكترونية.[26] يتراوح حجم جزيئات الفيروس بين 80-90 نانومتر. يظهر غلاف الفيروس في المجاهر الإلكترونية كزوج مميز من الأصداف الكثيفة الإلكترونية.[27]
يتكون الغلاف الفيروسي من طبقة ثنائية دهنية حيث يتم تثبيت غشاء (M) ، غلاف (E) وبروتينات الشوكة (S).[28] تزود بروتينات الشوكة الفيروس ببروزاته المنتفخة السطحية. يعد تفاعل بروتين الشوكة مع مستقبلات الخلية المضيفة التكميلية أمرًا محوريًا في تحديد الأنسجة الموجهةوالعدوى ونطاق الأنواع من الفيروس.[29][30]
داخل الغلاف ، يوجد قفيصة منواة ، الذي يتكون من نسخ متعددة من بروتين قفيصة المنواة (N) ، المرتبط بجينوم رنا (RNA) الأحادي الموجب (~ 30 زوج قاعدي) جينوم رنا في نمط خرزات مستمرة على خيط لتشكيل النمط..[31][32] يحمي غلاف الطبقة الدهنية وبروتينات الغشاء والقفيصة المنواة الفيروس عندما يكون خارج المضيف .[33]
دورة الحياة
يتبع الفيروس التاجي المرتبط بالسارس استراتيجية النسخ النموذجية لجميع الفيروسات التاجية.[19][34][35][36][37]
الالتصاق والدخول
يتم ربط ارتباط الفيروس التاجي المرتبط بالسارس(ڤيروس كورونا المرتبط بالمتلازمة التنفسية الحادة الوخيمة) بالخلية المضيفة بواسطة بروتين الشوكة ومستقبله.[38] يتعرف مجال ربط مستقبلات البروتين (RBD) على مستقبل الإنزيم المحول للأنجيوتنسين 2 (ACE2) ويرتبط به.[4] بعد الالتصاق، يمكن للفيروس دخول الخلية المضيفة من خلال مسارين مختلفين. يعتمد المسار الذي يتخذه الفيروس على الپروتياز المضيف المتاح لفك وتنشيط بروتين الشوكة المرتبط بالمستقبل..[39]
أول مسار يمكن أن يتخذه الفيروس التاجي للسارس للدخول إلى الخلية المضيفة هو التقام (ادخال خلوي) الخلايا وامتصاص الفيروس في الجسيم الداخلي . ثم يتم تنشيط البروتين الشوكة المستقبل من خلال المضيف تعتمد على درجة الحموضة سيستين بروتياز كاتيبسين L . يؤدي تنشيط بروتين الشوكة المرتبط بالمستقبل إلى حدوث تغير في الهيئة ، والاندماج اللاحق للغلاف الفيروسي مع جدار الجسيم الداخلي.[39]
بدلا من ذلك، يمكن للفيروس أن يدخل الخلية المضيفة مباشرة بواسطة التحلل البروتيني للمستقبل المرتبط به بروتين الشوكة من خلال المضيف TMPRSS2 أو TMPRSS11D سيرين بروتياز على سطح الخلية.[40][41] في الفيروس التاجي المرتبط بالسارس ، يؤدي تنشيط النهاية الكربوكسيلية لبروتين الشوكة إلى اندماج الغلاف الفيروسي مع غشاء الخلية المضيفة عن طريق إحداث تغييرات مطابقة غير مفهومة بالكامل.[42]
ترجمة الجينوم
| وظيفة فيروس كورونا بروتين غير هيكلي (nsps)[43] | |
|---|---|
| البروتين | الوظيفة |
| nsp1 | يعزز تدهور مرسال الحمض النووي الريبي للمضيف ، ويمنع ترجمة المضيف ؛
يمنع استجابة جهاز المناعة الفطري |
| nsp2 | يرتبط بالبروتينات المانعة ؛
وظيفة غير معروفة |
| nsp3 | بروتين غشائي متعدد النطاقات ؛ يتفاعل مع بروتين N ؛ يعزز التعبير السيتوكين . باباين يشق نطاق البروتين المتعدد ويثبط الاستجابة المناعية الفطرية pp1ab للمضيف. النطاقات الأخرى لها وظائف غير معروفة |
| nsp4 | بروتين سكافولد الغشائي
يسمح ببنية مناسبة للحويصلات الغشائية المزدوجة (DMVs) |
| nsp5 | 3CLPro يشق البروتين المتعدد pp1ab |
| nsp6 | بروتين سكافولد الغشائي
وظائف غير معروفة |
| nsp7 | تشكل مركب سداسي الشكل مع nsp8 ؛ مشبك العملية ل RdRp (nsp12) |
| nsp8 | تشكل مركب سداسي الشكل مع nsp7 ؛ المشبك العملية ل RdRp (nsp12) ؛ بمثابة بريمايز |
| nsp9 | بروتين ربط الرنا (RBP) |
| nsp10 | nsp16 و nsp14 عامل مساعد ؛ يشكل متغاير مع كليهما ؛ يحفز نشاط 2-O-MT (nsp16) و ExoN (nsp14) |
| nsp11 | وظيفة غير معروفة |
| nsp12 | بوليميراز الآر إن إيه المعتمد على الآر إن إيه (RdRp) |
| nsp13 | هيليكاز الرنا, 5′ ثلاثي الفوسفات |
| nsp14 | N7 ناقلة الميثيل ، 3′-5 ′ قاطع النيوكليوتيد الخارجي (اكسون) ؛ يضيف N7 MTase جينوم ExoN 5-غطاء ، يدقق الجينوم |
| nsp15 | قاطع النيوكليوتيد الداخلي (NendoU) |
| nsp16 | 2′-O-ناقل الميثيل (2-O-MT) ؛ يحمي الحمض الريبي النووي الفيروسي من MDA5 |
عد الاندماج ، يمر القفيصة المنواة في الهيولي ، حيث يتم إطلاق الجينوم الفيروسي.[38] يعمل الجينوم كمرسال الحمض النووي الريبي ، ويترجم الريبوسوم في الخلية ثلثي الجينوم ، الذي يتوافق مع إطار القراءة المفتوح ORF1a و ORF1b ، إلى بروتينين متراكبين كبيرين ، pp1a و pp1ab..
أكبر عديد البروتين pp1ab هو ناتج عن 1- تغير إطار ريبوزومي ناتج عن تسلسل زلق (UUUAAAC) و بنية حمض نووي ثانوية في نهاية إطار القراءة المفتوح ORF1a.[44] يسمح إطار الإطار الريبوسومي بالترجمة المستمرة لـ ORF1a متبوعًا بـ ORF1b.[45]
تحتوي البروتينات المتعددة على البروتياز الخاص بها ، PLpro و 3 CLpro ، اللذين يشقان البروتينات المتعددة في مواقع محددة مختلفة. ينتج انشقاق متعدد البروتين pp1ab 16 بروتينًا غير هيكلي (nsp1 إلى nsp16). تشتمل بروتينات المنتجة على بروتينات نسخ مختلفة مثل بوليميراز الآر إن إيه المعتمد على الآر إن إيه (RRP) ، هيليكاز الحمض النووي الريبي ، قاطع النيوكليوتيد الخارجي (إكسون).[46][47]).[45][36]
تناسخ و نسخ

يتحد عدد من بروتينات النسخ غير الهيكلي لتكوين مركب متعدد البروتين - نسخ- الاستنساخ (RTC)).[45] والبروتين الرئيسي للنسخ المتماثل هو بوليميراز الآر إن إيه المعتمد على الآر إن إيه (RdRp). وتشارك بشكل مباشر في التناسخ والنسخ من الرنا من شريط الرنا. تساعد البروتينات غير الهيكلية الأخرى في المركب في عملية النسخ والتناسخ.[43]
البروتين nsp15 هو 3'-5 'قاطع نيوكليوتيد خارجي الذي يوفر دقة إضافية لعملية التناسخ. يوفر قاطع نيوكليوتيد خارجي وظيفة التدقيق للمركب الذي تفتقر إليه بوليميراز الآر إن إيه المعتمد على الآر إن إيه. وبالمثل، البروتينات nsp7 وnsp8 تشكل شكل سداسي انزلاقي مشبكي كجزء من المركب الذي يزيد كثيرا من المعالجة من بوليميراز الآر إن إيه المعتمد على الآر إن إيه.[43] تتطلب الفيروسات التاجية زيادة الدقة والإنتاجية أثناء توليف الحمض النووي الريبي بسبب حجم الجينوم الكبير نسبيًا مقارنة بفيروسات الحمض النووي الريبي الأخرى.[48]
واحدة من الوظائف الرئيسية لمركب التناسخ المتماثل هي نسخ الجينوم الفيروسي. يتحكم بوليميراز الآر إن إيه المعتمد على الآر إن إيه مباشرة تخليق جزيئات رنا تحت الجينية السلبية الحسية من رنا الجينومي الإيجابي الحسي. ويتبع ذلك نسخ جزيئات رنا تحت الجينوم السلبية السلبية إلى مرسال الحمض النووي الريبي المقابلة لها بالمعنى الإيجابي.[49]
الوظيفة المهمة الأخرى لمركب النسخ المتماثل هي نسخ الجينوم الفيروسي. يتحكم بوليميراز الآر إن إيه المعتمد على الآر إن إيه مباشرة تخليق الحمض النووي الريبي الجيني السلبي من الحمض النووي الريبي الجيني الإيجابي. ويتبع ذلك تكرار الحمض النووي الريبي الجيني الإيجابي من الحمض النووي الريبي الجيني السلبي.[49]
يصبح الحمض النووي الريبي الجيني الإيجابي المنسوخ الجينوم لفيروسات النسل . مرسال الحمض النووي الريبي المختلف الأصغر هي نسخ من الثلث الأخير من جينوم الفيروس الذي يتبع إطارات القراءة ORF1a و ORF1b. تتم ترجمة هذه المرسالات الحمض النووي الريبي إلى البروتينات الهيكلية الأربعة (S و E و M و N) التي ستصبح جزءًا من جزيئات فيروس النسل وأيضاً ثمانية بروتينات ثانوية أخرى (orf3 إلى orf9b) التي تساعد الفيروس.[50]
التجمع والاطلاق
تحدث ترجمة الحمض النووي الريبي داخل الشبكة هيولية باطنة . تتحرك البروتينات الهيكلية الفيروسية S و E و M على طول المسار الإفرازي إلى حجرة جولجي المتوسطة . هناك ، توجه بروتينات M معظم التفاعلات بين البروتين والبروتين اللازمة لتجميع الفيروسات بعد ارتباطها بالقفيصة المنواة.[51]
يتم إطلاق فيروسات النسل من الخلية المضيفة عن طريق الايماس (اخراج خلوي) خلال الحويصلات الإفرازية.[51]
انظر ايضا
- SARS-like coronavirus WIV1 (SL-CoV-WIV1)
ملحوظة
- ^ The terms SARSr-CoV and SARS-CoV are sometimes used interchangeably, especially prior to the discovery of SARS-CoV-2.
المراجع
- ^ "ICTV Taxonomy history: Severe acute respiratory syndrome-related coronavirus" (html). International Committee on Taxonomy of Viruses (ICTV) (in الإنجليزية). Retrieved 27 January 2019.
- ^ Branswell, Helen (9 November 2015). "SARS-like virus in bats shows potential to infect humans, study finds". Stat News. Retrieved 20 February 2020.
{{cite news}}: Unknown parameter|name-list-format=ignored (|name-list-style=suggested) (help)CS1 maint: url-status (link) - ^ Wong AC, Li X, Lau SK, Woo PC (February 2019). "Global Epidemiology of Bat Coronaviruses". Viruses. 11 (2): 174. doi:10.3390/v11020174. PMC 6409556. PMID 30791586.
Most notably, horseshoe bats were found to be the reservoir of SARS-like CoVs, while palm civet cats are considered to be the intermediate host for SARS-CoVs [43,44,45].
{{cite journal}}: CS1 maint: unflagged free DOI (link) - ^ أ ب Ge XY, Li JL, Yang XL, Chmura AA, Zhu G, Epstein JH, et al. (November 2013). "Isolation and characterization of a bat SARS-like coronavirus that uses the ACE2 receptor". Nature. 503 (7477): 535–8. Bibcode:2013Natur.503..535G. doi:10.1038/nature12711. PMC 5389864. PMID 24172901.
- ^ "Virus Taxonomy: 2018 Release". International Committee on Taxonomy of Viruses (ICTV) (in الإنجليزية). October 2018. Retrieved 13 January 2019.
- ^ Woo PC, Huang Y, Lau SK, Yuen KY (August 2010). "Coronavirus genomics and bioinformatics analysis". Viruses. 2 (8): 1804–20. doi:10.3390/v2081803. PMC 3185738. PMID 21994708.
Figure 2. Phylogenetic analysis of RNA-dependent RNA polymerases (Pol) of coronaviruses with complete genome sequences available. The tree was constructed by the neighbor-joining method and rooted using Breda virus polyprotein.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - ^ أ ب ت "The species Severe acute respiratory syndrome-related coronavirus: classifying 2019-nCoV and naming it SARS-CoV-2". Nature Microbiology. March 2020. doi:10.1038/s41564-020-0695-z. PMID 32123347.
- ^ Lau SK, Li KS, Huang Y, Shek CT, Tse H, Wang M, et al. (March 2010). "Ecoepidemiology and complete genome comparison of different strains of severe acute respiratory syndrome-related Rhinolophus bat coronavirus in China reveal bats as a reservoir for acute, self-limiting infection that allows recombination events". Journal of Virology. 84 (6): 2808–19. doi:10.1128/JVI.02219-09. PMC 2826035. PMID 20071579.
- ^ Kieny, Marie-Paule. "After Ebola, a Blueprint Emerges to Jump-Start R&D". Scientific American Blog Network. Archived from the original on 20 December 2016. Retrieved 13 December 2016.
{{cite web}}: Unknown parameter|name-list-format=ignored (|name-list-style=suggested) (help) - ^ "LIST OF PATHOGENS". World Health Organization. Archived from the original on 20 December 2016. Retrieved 13 December 2016.
- ^ Wong AC, Li X, Lau SK, Woo PC (February 2019). "Global Epidemiology of Bat Coronaviruses". Viruses. 11 (2): 174. doi:10.3390/v11020174. PMC 6409556. PMID 30791586.
See Figure 1.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - ^ Woo PC, Huang Y, Lau SK, Yuen KY (August 2010). "Coronavirus genomics and bioinformatics analysis". Viruses. 2 (8): 1804–20. doi:10.3390/v2081803. PMC 3185738. PMID 21994708.
See Figure 1.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - ^ Woo PC, Huang Y, Lau SK, Yuen KY (August 2010). "Coronavirus genomics and bioinformatics analysis". Viruses. 2 (8): 1804–20. doi:10.3390/v2081803. PMC 3185738. PMID 21994708.
Furthermore, subsequent phylogenetic analysis using both complete genome sequence and proteomic approaches, it was concluded that SARSr-CoV is probably an early split-off from the Betacoronavirus lineage [1]; See Figure 2.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - ^ "Coronaviridae - Figures - Positive Sense RNA Viruses - Positive Sense RNA Viruses (2011)". International Committee on Taxonomy of Viruses (ICTV) (in الإنجليزية). Retrieved 2020-03-06.
See Figure 2.
- ^ Gouilh MA, Puechmaille SJ, Gonzalez JP, Teeling E, Kittayapong P, Manuguerra JC (October 2011). "SARS-Coronavirus ancestor's foot-prints in South-East Asian bat colonies and the refuge theory". Infection, Genetics and Evolution. 11 (7): 1690–702. doi:10.1016/j.meegid.2011.06.021. PMID 21763784.
Betacoronaviruses-b ancestors, meaning SARSr-CoVs ancestors, could have been historically hosted by the common ancestor of the Rhinolophidae and Hipposideridae and could have later evolved independently in the lineages leading towards Rhinolophidae and Hipposideridae betacoronaviruses.
- ^ Cui J, Han N, Streicker D, Li G, Tang X, Shi Z, et al. (October 2007). "Evolutionary relationships between bat coronaviruses and their hosts". Emerging Infectious Diseases. 13 (10): 1526–32. doi:10.3201/eid1310.070448. PMC 2851503. PMID 18258002.
- ^ Ge XY, Li JL, Yang XL, Chmura AA, Zhu G, Epstein JH, et al. (November 2013). "Isolation and characterization of a bat SARS-like coronavirus that uses the ACE2 receptor". Nature. 503 (7477): 535–8. Bibcode:2013Natur.503..535G. doi:10.1038/nature12711. PMC 5389864. PMID 24172901.
- ^ أ ب ت Snijder EJ, Bredenbeek PJ, Dobbe JC, Thiel V, Ziebuhr J, Poon LL, et al. (August 2003). "Unique and conserved features of genome and proteome of SARS-coronavirus, an early split-off from the coronavirus group 2 lineage". Journal of Molecular Biology. 331 (5): 991–1004. doi:10.1016/S0022-2836(03)00865-9. PMID 12927536.
The SARS-CoV genome is ∼29.7 kb long and contains 14 open reading frames (ORFs) flanked by 5′ and 3′-untranslated regions of 265 and 342 nucleotides, respectively (Figure 1).
- ^ أ ب Fehr AR, Perlman S (2015). Maier HJ, Bickerton E, Britton P (eds.). "Coronaviruses: an overview of their replication and pathogenesis". Methods in Molecular Biology. 1282. Springer: 1–23. doi:10.1007/978-1-4939-2438-7_1. ISBN 978-1-4939-2438-7. PMC 4369385. PMID 25720466.
{{cite journal}}: Cite journal requires|journal=(help); Unknown parameter|name-list-format=ignored (|name-list-style=suggested) (help) - ^ Fehr AR, Perlman S (2015). Maier HJ, Bickerton E, Britton P (eds.). "An Overview of Their Replication and Pathogenesis; Section 2 Genomic Organization". Methods in Molecular Biology. Springer. 1282: 1–23. doi:10.1007/978-1-4939-2438-7_1. ISBN 978-1-4939-2438-7. PMC 4369385. PMID 25720466.
{{cite journal}}: Unknown parameter|name-list-format=ignored (|name-list-style=suggested) (help) - ^ McBride R, Fielding BC (November 2012). "The role of severe acute respiratory syndrome (SARS)-coronavirus accessory proteins in virus pathogenesis". Viruses. 4 (11): 2902–23. doi:10.3390/v4112902. PMC 3509677. PMID 23202509.
See Table 1.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - ^ Tang X, Li G, Vasilakis N, Zhang Y, Shi Z, Zhong Y, Wang LF, Zhang S (March 2009). "Differential stepwise evolution of SARS coronavirus functional proteins in different host species". BMC Evolutionary Biology. 9: 52. doi:10.1186/1471-2148-9-52. PMC 2676248. PMID 19261195.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - ^ Narayanan, Krishna; Huang, Cheng; Makino, Shinji (April 2008). "SARS coronavirus Accessory Proteins". Virus research. 133 (1): 113–121. doi:10.1016/j.virusres.2007.10.009. ISSN 0168-1702. PMC 2720074. PMID 18045721.
See Table 1.
- ^ أ ب McBride R, Fielding BC (November 2012). "The role of severe acute respiratory syndrome (SARS)-coronavirus accessory proteins in virus pathogenesis". Viruses. 4 (11): 2902–23. doi:10.3390/v4112902. PMC 3509677. PMID 23202509.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - ^ Snijder EJ, Bredenbeek PJ, Dobbe JC, Thiel V, Ziebuhr J, Poon LL, et al. (August 2003). "Unique and conserved features of genome and proteome of SARS-coronavirus, an early split-off from the coronavirus group 2 lineage". Journal of Molecular Biology. 331 (5): 991–1004. doi:10.1016/S0022-2836(03)00865-9. PMID 12927536.
See Figure 1.
- ^ Goldsmith CS, Tatti KM, Ksiazek TG, Rollin PE, Comer JA, Lee WW, et al. (February 2004). "Ultrastructural characterization of SARS coronavirus". Emerging Infectious Diseases. 10 (2): 320–6. doi:10.3201/eid1002.030913. PMC 3322934. PMID 15030705.
Virions acquired an envelope by budding into the cisternae and formed mostly spherical, sometimes pleomorphic, particles that averaged 78 nm in diameter (Figure 1A).
- ^ Neuman BW, Adair BD, Yoshioka C, Quispe JD, Orca G, Kuhn P, et al. (August 2006). "Supramolecular architecture of severe acute respiratory syndrome coronavirus revealed by electron cryomicroscopy". Journal of Virology. 80 (16): 7918–28. doi:10.1128/JVI.00645-06. PMC 1563832. PMID 16873249.
Particle diameters ranged from 50 to 150 nm, excluding the spikes, with mean particle diameters of 82 to 94 nm; Also See Figure 1 for double shell.
- ^ Lai MM, Cavanagh D (1997). "The molecular biology of coronaviruses". Advances in Virus Research. 48: 1–100. doi:10.1016/S0065-3527(08)60286-9. PMID 9233431.
- ^ Masters PS (2006-01-01). "The molecular biology of coronaviruses". Advances in Virus Research. Advances in Virus Research. Academic Press. 66: 193–292. doi:10.1016/S0065-3527(06)66005-3. ISBN 9780120398690. PMID 16877062.
Nevertheless, the interaction between S protein and receptor remains the principal, if not sole, determinant of coronavirus host species range and tissue tropism.
- ^ Cui J, Li F, Shi ZL (March 2019). "Origin and evolution of pathogenic coronaviruses". Nature Reviews. Microbiology. 17 (3): 181–192. doi:10.1038/s41579-018-0118-9. PMID 30531947.
Different SARS-CoV strains isolated from several hosts vary in their binding affinities for human ACE2 and consequently in their infectivity of human cells76,78 (Fig. 6b)
- ^ Fehr AR, Perlman S (2015). Maier HJ, Bickerton E, Britton P (eds.). "An Overview of Their Replication and Pathogenesis; Section 2 Genomic Organization". Methods in Molecular Biology. Springer. 1282: 1–23. doi:10.1007/978-1-4939-2438-7_1. ISBN 978-1-4939-2438-7. PMC 4369385. PMID 25720466.
See section: Virion Structure.
{{cite journal}}: Unknown parameter|name-list-format=ignored (|name-list-style=suggested) (help) - ^ Chang CK, Hou MH, Chang CF, Hsiao CD, Huang TH (March 2014). "The SARS coronavirus nucleocapsid protein--forms and functions". Antiviral Research. 103: 39–50. doi:10.1016/j.antiviral.2013.12.009. PMID 24418573.
See Figure 4c.
- ^ Neuman BW, Kiss G, Kunding AH, Bhella D, Baksh MF, Connelly S, et al. (April 2011). "A structural analysis of M protein in coronavirus assembly and morphology". Journal of Structural Biology. 174 (1): 11–22. doi:10.1016/j.jsb.2010.11.021. PMC 4486061. PMID 21130884.
See Figure 10.
- ^ Tok TT, Tatar G. "Structures and Functions of Coronavirus Proteins: Molecular Modeling of Viral Nucleoprotein" (PDF).
- ^ Lal, Sunil K, ed. (2010). Molecular Biology of the SARS-Coronavirus. doi:10.1007/978-3-642-03683-5. ISBN 978-3-642-03682-8.
{{cite book}}: Unknown parameter|name-list-format=ignored (|name-list-style=suggested) (help) - ^ أ ب Cui H, Gao Z, Liu M, Lu S, Mkandawire W, Mo S, Narykov O, Srinivasan S, Korkin D (January 2020). "Structural genomics and interactomics of 2019 Wuhan novel coronavirus, 2019-nCoV, indicate evolutionary conserved functional regions of viral proteins". bioRxiv. doi:10.1101/2020.02.10.942136.
- ^ Wu F, Zhao S, Yu B, Chen YM, Wang W, Hu Y, et al. (January 2020). "Complete genome characterisation of a novel coronavirus associated with severe human respiratory disease in Wuhan, China". bioRxiv. doi:10.1101/2020.01.24.919183.
- ^ أ ب Fehr AR, Perlman S (2015). Maier HJ, Bickerton E, Britton P (eds.). "Coronaviruses: an overview of their replication and pathogenesis". Methods in Molecular Biology. 1282. Springer: 1–23. doi:10.1007/978-1-4939-2438-7_1. ISBN 978-1-4939-2438-7. PMC 4369385. PMID 25720466.
See section: Coronavirus Life Cycle – Attachment and Entry
{{cite journal}}: Cite journal requires|journal=(help); Unknown parameter|name-list-format=ignored (|name-list-style=suggested) (help) - ^ أ ب Simmons G, Zmora P, Gierer S, Heurich A, Pöhlmann S (December 2013). "Proteolytic activation of the SARS-coronavirus spike protein: cutting enzymes at the cutting edge of antiviral research". Antiviral Research. 100 (3): 605–14. doi:10.1016/j.antiviral.2013.09.028. PMC 3889862. PMID 24121034.
See Figure 2.
- ^ Heurich A, Hofmann-Winkler H, Gierer S, Liepold T, Jahn O, Pöhlmann S (January 2014). "TMPRSS2 and ADAM17 cleave ACE2 differentially and only proteolysis by TMPRSS2 augments entry driven by the severe acute respiratory syndrome coronavirus spike protein". Journal of Virology. 88 (2): 1293–307. doi:10.1128/JVI.02202-13. PMC 3911672. PMID 24227843.
The SARS-CoV can hijack two cellular proteolytic systems to ensure the adequate processing of its S protein. Cleavage of SARS-S can be facilitated by cathepsin L, a pH-dependent endo-/lysosomal host cell protease, upon uptake of virions into target cell endosomes (25). Alternatively, the type II transmembrane serine proteases (TTSPs) TMPRSS2 and HAT can activate SARS-S, presumably by cleavage of SARS-S at or close to the cell surface, and activation of SARS-S by TMPRSS2 allows for cathepsin L-independent cellular entry (26,–28).
- ^ Zumla A, Chan JF, Azhar EI, Hui DS, Yuen KY (May 2016). "Coronaviruses - drug discovery and therapeutic options". Nature Reviews. Drug Discovery. 15 (5): 327–47. doi:10.1038/nrd.2015.37. PMID 26868298.
S is activated and cleaved into the S1 and S2 subunits by other host proteases, such as transmembrane protease serine 2 (TMPRSS2) and TMPRSS11D, which enables cell surface non-endosomal virus entry at the plasma membrane.
- ^ Li Z, Tomlinson AC, Wong AH, Zhou D, Desforges M, Talbot PJ, et al. (October 2019). "The human coronavirus HCoV-229E S-protein structure and receptor binding". eLife. 8. doi:10.7554/eLife.51230. PMC 6970540. PMID 31650956.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - ^ أ ب ت Fehr AR, Perlman S (2015). Maier HJ, Bickerton E, Britton P (eds.). "Coronaviruses: an overview of their replication and pathogenesis". Methods in Molecular Biology. 1282. Springer: 1–23. doi:10.1007/978-1-4939-2438-7_1. ISBN 978-1-4939-2438-7. PMC 4369385. PMID 25720466.
See Table 2.
{{cite journal}}: Cite journal requires|journal=(help); Unknown parameter|name-list-format=ignored (|name-list-style=suggested) (help) - ^ Masters PS (2006-01-01). "The molecular biology of coronaviruses". Advances in Virus Research. Academic Press. 66: 193–292. doi:10.1016/S0065-3527(06)66005-3. ISBN 9780120398690. PMID 16877062.
See Figure 8.
- ^ أ ب ت Fehr AR, Perlman S (2015). Maier HJ, Bickerton E, Britton P (eds.). "Coronaviruses: an overview of their replication and pathogenesis". Methods in Molecular Biology. 1282. Springer: 1–23. doi:10.1007/978-1-4939-2438-7_1. ISBN 978-1-4939-2438-7. PMC 4369385. PMID 25720466.
See section: Replicase Protein Expression
{{cite journal}}: Cite journal requires|journal=(help); Unknown parameter|name-list-format=ignored (|name-list-style=suggested) (help) - ^
{{cite book}}: Empty citation (help) - ^ قالب:استشهاد بدورية أكاديمية
- ^ Sexton NR, Smith EC, Blanc H, Vignuzzi M, Peersen OB, Denison MR (August 2016). "Homology-Based Identification of a Mutation in the Coronavirus RNA-Dependent RNA Polymerase That Confers Resistance to Multiple Mutagens". Journal of Virology. 90 (16): 7415–28. doi:10.1128/JVI.00080-16. PMC 4984655. PMID 27279608.
Finally, these results, combined with those from previous work (33, 44), suggest that CoVs encode at least three proteins involved in fidelity (nsp12-RdRp, nsp14-ExoN, and nsp10), supporting the assembly of a multiprotein replicase-fidelity complex, as described previously (38).
- ^ أ ب Fehr AR, Perlman S (2015). Maier HJ, Bickerton E, Britton P (eds.). "Coronaviruses: an overview of their replication and pathogenesis". Methods in Molecular Biology. 1282. Springer: 1–23. doi:10.1007/978-1-4939-2438-7_1. ISBN 978-1-4939-2438-7. PMC 4369385. PMID 25720466.
See section: Corona Life Cycle – Replication and Transcription
{{cite journal}}: Cite journal requires|journal=(help); Unknown parameter|name-list-format=ignored (|name-list-style=suggested) (help) - ^ Fehr AR, Perlman S (2015). Maier HJ, Bickerton E, Britton P (eds.). "Coronaviruses: an overview of their replication and pathogenesis". Methods in Molecular Biology. 1282. Springer: 1–23. doi:10.1007/978-1-4939-2438-7_1. ISBN 978-1-4939-2438-7. PMC 4369385. PMID 25720466.
See Figure 1.
{{cite journal}}: Cite journal requires|journal=(help); Unknown parameter|name-list-format=ignored (|name-list-style=suggested) (help) - ^ أ ب Fehr AR, Perlman S (2015). Maier HJ, Bickerton E, Britton P (eds.). "Coronaviruses: an overview of their replication and pathogenesis". Methods in Molecular Biology. 1282. Springer: 1–23. doi:10.1007/978-1-4939-2438-7_1. ISBN 978-1-4939-2438-7. PMC 4369385. PMID 25720466.
See section: Coronavirus Life Cycle – Assembly and Release
{{cite journal}}: Cite journal requires|journal=(help); Unknown parameter|name-list-format=ignored (|name-list-style=suggested) (help)
Further reading
- Peiris JS, Lai ST, Poon LL, Guan Y, Yam LY, Lim W, et al. (April 2003). "Coronavirus as a possible cause of severe acute respiratory syndrome". Lancet. 361 (9366): 1319–25. doi:10.1016/s0140-6736(03)13077-2. PMID 12711465.
- Rota PA, Oberste MS, Monroe SS, Nix WA, Campagnoli R, Icenogle JP, et al. (May 2003). "Characterization of a novel coronavirus associated with severe acute respiratory syndrome". Science. 300 (5624): 1394–9. Bibcode:2003Sci...300.1394R. doi:10.1126/science.1085952. PMID 12730500.
- Marra MA, Jones SJ, Astell CR, Holt RA, Brooks-Wilson A, Butterfield YS, et al. (May 2003). "The Genome sequence of the SARS-associated coronavirus". Science. 300 (5624): 1399–404. Bibcode:2003Sci...300.1399M. doi:10.1126/science.1085953. PMID 12730501.
- Snijder EJ, Bredenbeek PJ, Dobbe JC, Thiel V, Ziebuhr J, Poon LL, et al. (August 2003). "Unique and conserved features of genome and proteome of SARS-coronavirus, an early split-off from the coronavirus group 2 lineage". Journal of Molecular Biology. 331 (5): 991–1004. CiteSeerX 10.1.1.319.7007. doi:10.1016/S0022-2836(03)00865-9. PMID 12927536.
- Yount B, Roberts RS, Lindesmith L, Baric RS (August 2006). "Rewiring the severe acute respiratory syndrome coronavirus (SARS-CoV) transcription circuit: engineering a recombination-resistant genome". Proceedings of the National Academy of Sciences of the United States of America. 103 (33): 12546–51. Bibcode:2006PNAS..10312546Y. doi:10.1073/pnas.0605438103. PMC 1531645. PMID 16891412.
- Thiel V, ed. (2007). Coronaviruses: Molecular and Cellular Biology (1st ed.). Caister Academic Press. ISBN 978-1-904455-16-5.
- Enjuanes L, Sola I, Zúñiga S, Almazán F (2008). "Coronavirus Replication and Interaction with Host". In Mettenleiter TC, Sobrino F (eds.). Animal Viruses: Molecular Biology. Caister Academic Press. ISBN 978-1-904455-22-6.
وصلات خارجية
- WHO press release identifying and naming the SARS virus
- The SARS virus genetic map
- Science special on the SARS virus (free content: no registration required)
- McGill University SARS Resources at the Wayback Machine (archived مارس 1, 2005)
- U.S. Centers for Disease Control and Prevention (CDC) SARS home
- World Health Organization on alert



