تطور موجه
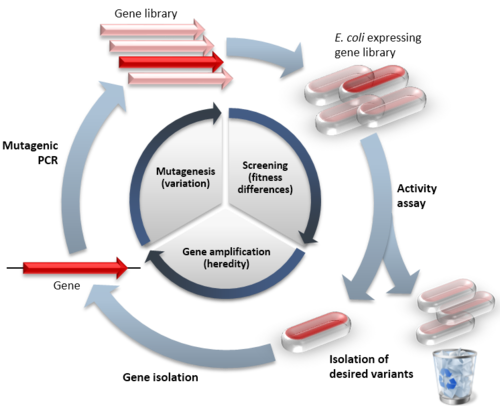
التطور الموجه Directed evolution، هي طريقة تستخدم هندسة الپروتين لمحاكاة عملية الانتخاب الطبيعي لتوجيه الپروتينات أو الأحماض النووية إلى هدف يحدده المستخدم.[1] يتألف التطور الموجه من تعريض حين معين لدورات متكررة من التطفير (خلق مكتبة متغيرتات)، اختيار (التعبير عن المتغيرات وعزل الافراد حسب الوظيفة المرغوبة)، وتضخيم (توليد قالب للجولة التالية). يمكن أداء هذه العملية على الخلايا الحية أو في المعمل. يستخدم التطور الموجه في هندسة الپروتين كبديل للپروتينات المعدلة منطقياً، ولدراسات المبادئ التطورية الأساسية في بيئة معملية ومسيطر عليها.
التاريخ
التطور المبكر لتقنية التطور الموجه لهندسة الپروتين كان بقيادة فرانسس أرنولد، التي حازت نصف جائزة نوبل في الفيزياء 2018[2]. بناءاً على العمل المبكر على هندسة الإنزيمات من أجل الاستقرار في المحاليل العضوية، قام تشن وأرنولد[3] بتطبيق التطفير العشوائي المجمع مع تفحص نشاط الانزيمات في محلول عضوي لتحديد الطفرات المناسبة. سرعان ما توسع استخدام التطور الموجه كتقنية في أوائل التسعينيات مع تطور التقنيات الجديدة لتعديل وفحص الجينات.
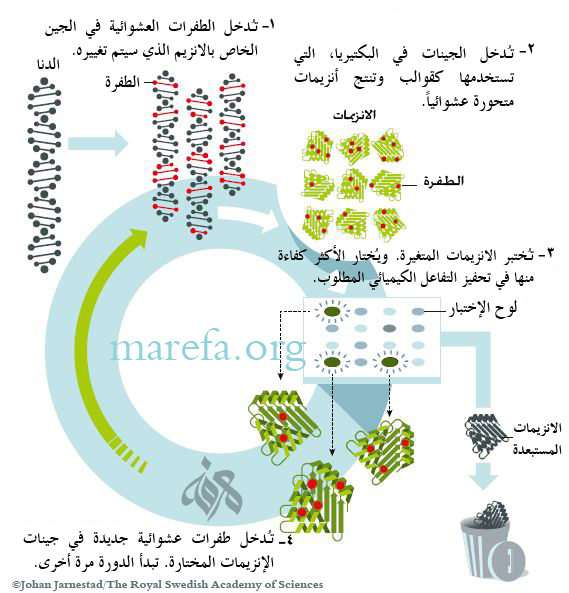
المبادئ
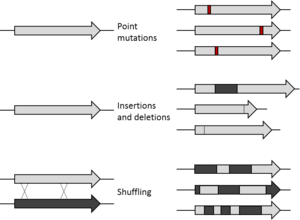
توليد التباين
الكشف عن فرق التطابق
الاختيار
العرض
ضمان الصفات الوراثية

مقارنة بتصميم الپروتين العقلاني
مميزات التطور الموجه
قيود التطور الموجه
الاستخدامات
هندسة الپروتين
هندسة الپروتين كأداة، كان التطور الموجه أكثر نجاحاً في المجالات الثلاث التالية: As a protein engineering tool, DE has been most successful in three areas:
- تحسين استقرار الپروتين للاستخدام التقني الحيوي عند درجات الحرارة المرتفعة أو في المذيبات الصعبة.[4][5]
- تعزيز قابلية ارتباط الأجسام المضادة العلاجية (ألفة النضج)[6] ونشاط الإنزيمات المعاد تصميمها.[7]
- تبديل خاصية التركيز للانزيمات القائمة،[8][9][10][11] (عادة ما تستخدم في الصناعة).[12]
دراسات التطور
دراسة التطور الطبيعي تعتمد تقليدياً على العضيات القائمة وجيناتها. ومع ذلك، فإن الأبحاث محدودة بشكل أساسي لنقص الأحفورات (وخاصة نقص تسلسلات الدنا القديم)[13][14] والمعرفة غير الكاملة بالظروف البيئية القديمة. يتحرى التطور الموجه التطور في أنظمة الجينات المتحكم بها للانزيمات الفردية،[15][16][17] انزيم الحمض الريبي النووي[18] والنسخ المتماثلة[19][20] (المماثل للتطور التجريبي لحقيقيات النوى،[21][22] بدائيات النوى[23] والڤيروسات[24]).
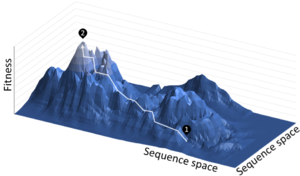
يسمح التطور الموجه بالتحكم في بالضغط التطوري، معدل الطفرات والبيئة (سواء البيئة اللاأحيائية مثل درجة الحرارة، أو البيئية الأحيائية، مثل الجينات الأخرى الموجودة في العضيات). بالإضافة إلى ذلك، هناك سجل كامل لجميع الجينات التطورية. يسمح هذا بإجراء قياسات مفصلة للعمليات التطورية، على سبيل المثال، الروكبة (كبت صفات جين بواسطة جين آخر epistasis)، التطورية، القيد التكيفي[25][26] fitness landscapes،[27] والشبكات العصبية.[28]
انظر أيضاً
- التطبيقات:
- التطفير:
- الاختيار والفحص:
- شركات توفر خدمات التطور الموجه للعملاء
المصادر
- ^ Lutz, S (Dec 2010). "Beyond directed evolution--semi-rational protein engineering and design". Curr Opin Biotechnol. 21: 734–43. doi:10.1016/j.copbio.2010.08.011. PMC 2982887. PMID 20869867.
- ^ "The Nobel Prize in Chemistry 2018". NobelPrize.org (in الإنجليزية الأمريكية). Retrieved 2018-10-03.
- ^ Chen, Keqin; Arnold, Frances H. (1991). "Enzyme Engineering for Nonaqueous Solvents: Random Mutagenesis to Enhance Activity of Subtilisin E in Polar Organic Media". Bio/Technology (in الإنجليزية). 9 (11): 1073–1077. doi:10.1038/nbt1191-1073. ISSN 0733-222X.
- ^ Gatti-Lafranconi, P; Natalello, A; Rehm, S; Doglia, SM; Pleiss, J; Lotti, M (8 January 2010). "Evolution of stability in a cold-active enzyme elicits specificity relaxation and highlights substrate-related effects on temperature adaptation". Journal of Molecular Biology. 395 (1): 155–66. doi:10.1016/j.jmb.2009.10.026. PMID 19850050.
- ^ Zhao, H; Arnold, FH (January 1999). "Directed evolution converts subtilisin E into a functional equivalent of thermitase". Protein engineering. 12 (1): 47–53. doi:10.1093/protein/12.1.47. PMID 10065710.
- ^ Hawkins, RE; Russell, SJ; Winter, G (5 August 1992). "Selection of phage antibodies by binding affinity. Mimicking affinity maturation". Journal of Molecular Biology. 226 (3): 889–96. doi:10.1016/0022-2836(92)90639-2. PMID 1507232.
- ^ Giger, L; Caner, S; Obexer, R; Kast, P; Baker, D; Ban, N; Hilvert, D (August 2013). "Evolution of a designed retro-aldolase leads to complete active site remodeling". Nature Chemical Biology. 9 (8): 494–8. doi:10.1038/nchembio.1276. PMC 3720730. PMID 23748672.
- ^ Shaikh, FA; Withers, SG (April 2008). "Teaching old enzymes new tricks: engineering and evolution of glycosidases and glycosyl transferases for improved glycoside synthesis". Biochemistry and cell biology = Biochimie et biologie cellulaire. 86 (2): 169–77. doi:10.1139/o07-149. PMID 18443630.
- ^ Cheriyan, M; Walters, MJ; Kang, BD; Anzaldi, LL; Toone, EJ; Fierke, CA (1 November 2011). "Directed evolution of a pyruvate aldolase to recognize a long chain acyl substrate". Bioorganic & Medicinal Chemistry. 19 (21): 6447–53. doi:10.1016/j.bmc.2011.08.056. PMC 3209416. PMID 21944547.
- ^ MacBeath, G; Kast, P; Hilvert, D (20 March 1998). "Redesigning enzyme topology by directed evolution". Science. 279 (5358): 1958–61. doi:10.1126/science.279.5358.1958. PMID 9506949.
- ^ Toscano, MD; Woycechowsky, KJ; Hilvert, D (2007). "Minimalist active-site redesign: teaching old enzymes new tricks". Angewandte Chemie International Edition in English. 46 (18): 3212–36. doi:10.1002/anie.200604205. PMID 17450624.
- ^ Turner, NJ (August 2009). "Directed evolution drives the next generation of biocatalysts". Nature Chemical Biology. 5 (8): 567–73. doi:10.1038/nchembio.203. PMID 19620998.
- ^ Pääbo, S; Poinar, H; Serre, D; Jaenicke-Despres, V; Hebler, J; Rohland, N; Kuch, M; Krause, J; Vigilant, L; Hofreiter, M (2004). "Genetic analyses from ancient DNA". Annual Review of Genetics. 38 (1): 645–79. doi:10.1146/annurev.genet.37.110801.143214. PMID 15568989.
- ^ Höss, M; Jaruga, P; Zastawny, TH; Dizdaroglu, M; Pääbo, S (1 April 1996). "DNA damage and DNA sequence retrieval from ancient tissues". Nucleic Acids Research. 24 (7): 1304–7. doi:10.1093/nar/24.7.1304. PMC 145783. PMID 8614634.
- ^ Bloom, JD; Arnold, FH (16 June 2009). "In the light of directed evolution: pathways of adaptive protein evolution". Proceedings of the National Academy of Sciences of the United States of America. 106 Suppl 1 (Supplement_1): 9995–10000. doi:10.1073/pnas.0901522106. PMC 2702793. PMID 19528653.
- ^ Moses, AM; Davidson, AR (17 May 2011). "In vitro evolution goes deep". Proceedings of the National Academy of Sciences of the United States of America. 108 (20): 8071–2. doi:10.1073/pnas.1104843108. PMC 3100951. PMID 21551096.
- ^ Goldsmith, M; Tawfik, DS (August 2012). "Directed enzyme evolution: beyond the low-hanging fruit". Current Opinion in Structural Biology. 22 (4): 406–12. doi:10.1016/j.sbi.2012.03.010. PMID 22579412.
- ^ Salehi-Ashtiani, K; Szostak, JW (1 November 2001). "In vitro evolution suggests multiple origins for the hammerhead ribozyme". Nature. 414 (6859): 82–4. doi:10.1038/35102081. PMID 11689947.
- ^ Sumper, M; Luce, R (January 1975). "Evidence for de novo production of self-replicating and environmentally adapted RNA structures by bacteriophage Qbeta replicase". Proceedings of the National Academy of Sciences of the United States of America. 72 (1): 162–6. doi:10.1073/pnas.72.1.162. PMC 432262. PMID 1054493.
- ^ Mills, DR; Peterson, RL; Spiegelman, S (July 1967). "An extracellular Darwinian experiment with a self-duplicating nucleic acid molecule". Proceedings of the National Academy of Sciences of the United States of America. 58 (1): 217–24. doi:10.1073/pnas.58.1.217. PMC 335620. PMID 5231602.
- ^ Marden, JH; Wolf, MR; Weber, KE (November 1997). "Aerial performance of Drosophila melanogaster from populations selected for upwind flight ability". The Journal of Experimental Biology. 200 (Pt 21): 2747–55. PMID 9418031.
- ^ Ratcliff, WC; Denison, RF; Borrello, M; Travisano, M (31 January 2012). "Experimental evolution of multicellularity". Proceedings of the National Academy of Sciences of the United States of America. 109 (5): 1595–600. doi:10.1073/pnas.1115323109. PMC 3277146. PMID 22307617.
- ^ Barrick, JE; Yu, DS; Yoon, SH; Jeong, H; Oh, TK; Schneider, D; Lenski, RE; Kim, JF (29 October 2009). "Genome evolution and adaptation in a long-term experiment with Escherichia coli". Nature. 461 (7268): 1243–7. doi:10.1038/nature08480. PMID 19838166.
- ^ Heineman, RH; Molineux, IJ; Bull, JJ (August 2005). "Evolutionary robustness of an optimal phenotype: re-evolution of lysis in a bacteriophage deleted for its lysin gene". Journal of Molecular Evolution. 61 (2): 181–91. doi:10.1007/s00239-004-0304-4. PMID 16096681.
- ^ Steinberg, Barrett; Ostermeier, Marc (2016-01-01). "Environmental changes bridge evolutionary valleys". Science Advances (in الإنجليزية). 2 (1): e1500921. doi:10.1126/sciadv.1500921. ISSN 2375-2548. PMC 4737206. PMID 26844293.
- ^ Arnold, FH; Wintrode, PL; Miyazaki, K; Gershenson, A (February 2001). "How enzymes adapt: lessons from directed evolution". Trends in Biochemical Sciences. 26 (2): 100–6. doi:10.1016/s0968-0004(00)01755-2. PMID 11166567.
- ^ Aita, T; Hamamatsu, N; Nomiya, Y; Uchiyama, H; Shibanaka, Y; Husimi, Y (5 July 2002). "Surveying a local fitness landscape of a protein with epistatic sites for the study of directed evolution". Biopolymers. 64 (2): 95–105. doi:10.1002/bip.10126. PMID 11979520.
- ^ Bloom, JD; Raval, A; Wilke, CO (January 2007). "Thermodynamics of neutral protein evolution". Genetics. 175 (1): 255–66. doi:10.1534/genetics.106.061754. PMC 1775007. PMID 17110496.
وصلات خارجية
- مجموعات بحثية
- The Dan Tawfik Research Group
- The Ulrich Schwaneberg Research Group
- The Frances Arnold Research Group
- The Huimin Zhao Research Group
- The Manfred Reetz Research Group
- The Donald Hilvert Group
- The Darren Hart Research Group
- The Chang Liu Research Group
- The David Liu Research Group
- The Douglas Clark Research Group
- The Paul Dalby Research Group
- SeSaM-Biotech - Directed Evolution
- Prof. Reetz explains the principle of Directed Evolution
- Codexis, Inc.