تنادد تسلسل

التسلسلات هي الأحماض الأمينية لبقايا 120-180 پروتين. البقايا المحفوظة عبر جميع التسلسلات ملونة بالرمادي. أسفل تسلسلات الپروتين يوجد مفتاح يشير إلى التسلسل المحفوظ (*)، conservative mutation (.)، وnon-conservative mutations ( ).[1]
تنادد التسلسل Sequence homology، هو تنادد بين الپروتين وتسلسلات الدنا، والمعرفة من حيث السلف المشترك في التاريخ التطوري للحياة. قطاعان من الدان يمكنهم أن يتشاركا سلف مشترك إما بسبب حدث الانتواع، أو بسبب حدث النسخ.[2]
التنادد بين الپروتينات أو الدنا يتم استنتاجه عادة من تشابه تسلسلها. يمثل التشابه الواضح دليلاً قوياً على أن هناك تسلسلين مرتبطين عن طريق التطور المتباعد من السلف المشترك. تستخدم تراصفات التسلسلات المتعددة للإشارة إلى إلى مناطق تنادد كل تسلسل.
عادة ما يستخدم مصطلح "تنادد النسبة المئوية" بمعنى "تشابه التسلسل". النسبة المئوية للبقايا المتطابقة (تطابق النسبة المئوية) أو النسبة المئوية للبقايا محفوظة مع الخصائص الفيزيوكيميائية المتشابه (تشابه النسبة المئوية)، على سبيل المثال الليوسين والآيزوليوسين، عادة ما تستخدم "لتحديد مقدار التنادد". تبعاً لتعريف التنادد الموضح أعلاه، فإن هذا المصطلح غير صحيح حيث أن تشابه التسلسل هو الرصد، بينما التنادد هو النتيجة. تكون التسلسلات إما متناددة أو غير متناددة. كما في حالة البنى التشريحية، قد يحدث تشابه التسلسل المرتفع بسبب التطور التقاربي، أو، كما في حالة التسلسلات الأقصر، بمحض الصدفة، مما يعني أنها ليست تناددية. مناطق التسلسل المتنادد تسمى أيضاً المحفوظة. يجب عدم الخلط بينها وبين الحفظ في تسلسلات الحمض الأميني، حيث يكون الحمض الأميني في موضع محدد تم استبداله بآخر له نفس الخصائص الفيزيوكيميائية المكافئة وظيفياً.
قد يحدث تنادد جزئي عندما يتشارك قطاع في التسلسل المقارن أصل مشترك، بينما لا تفعل بقية القطاعات. قد ينتج هذا التنادد الجزئي عن حدث انصهار جيني.
الانتواع
قاعدة بيانات جينات الانتواع
- eggNOG[3]
- InParanoid[4] focuses on pairwise ortholog relationships
- OrthoDB[5] appreciates that the orthology concept is relative to different speciation points by providing a hierarchy of orthologs along the species tree.
- OrthoMCL[6]
- OMA
- Roundup[7]
- OrthoMaM[8] for mammals
- OrthologID[9]
- GreenPhylDB[10] للنباتات
التماثل التشريحي
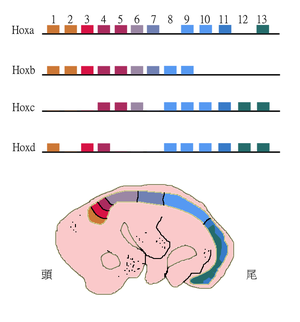
المناطق المتماثلة تشريحياً
نسخ الجينوم الكامل
نقل الجين الأفقي
Gametology
انظر أيضاً
- تنادد عميق
- EggNOG (قاعدة بيانات)
- OrthoDB
- Orthologous MAtrix (OMA)
- عائلة الپروتين
- العائلة العظمى للپروتينات
- TreeFam
- Syntelog
المصادر
- ^ "Clustal FAQ #Symbols". Clustal. Retrieved 8 December 2014.
- ^ Koonin EV (2005). "Orthologs, paralogs, and evolutionary genomics". Annual Review of Genetics. 39: 309–38. doi:10.1146/annurev.genet.39.073003.114725. PMID 16285863.
- ^ eggNOG: evolutionary genealogy of genes: Non-supervised Orthologous Groups
Muller J, Szklarczyk D, Julien P, et al. (January 2010). "eggNOG v2.0: extending the evolutionary genealogy of genes with enhanced non-supervised orthologous groups, species and functional annotations". Nucleic Acids Research. 38 (Database issue): D190–5. doi:10.1093/nar/gkp951. PMC 2808932. PMID 19900971. - ^ Inparanoid: Eukaryotic Ortholog Groups
Ostlund G, Schmitt T, Forslund K, et al. (January 2010). "InParanoid 7: new algorithms and tools for eukaryotic orthology analysis". Nucleic Acids Research. 38 (Database issue): D196–203. doi:10.1093/nar/gkp931. PMC 2808972. PMID 19892828. - ^ Zdobnov, EM; Tegenfeldt, F; Kuznetsov, D; Waterhouse, RM; Simão, FA; Ioannidis, P; Seppey, M; Loetscher, A; Kriventseva, EV (28 November 2016). "OrthoDB v9.1: cataloging evolutionary and functional annotations for animal, fungal, plant, archaeal, bacterial and viral orthologs". Nucleic Acids Research. 45: D744–D749. doi:10.1093/nar/gkw1119. PMID 27899580.
- ^ OrthoMCL: Identification of Ortholog Groups for Eukaryotic Genomes
Chen F, Mackey AJ, Stoeckert CJ, Roos DS (January 2006). "OrthoMCL-DB: querying a comprehensive multi-species collection of ortholog groups". Nucleic Acids Research. 34 (Database issue): D363–8. doi:10.1093/nar/gkj123. PMC 1347485. PMID 16381887. - ^ Roundup
Deluca TF, Wu IH, Pu J, et al. (August 2006). "Roundup: a multi-genome repository of orthologs and evolutionary distances". Bioinformatics. 22 (16): 2044–6. doi:10.1093/bioinformatics/btl286. PMID 16777906. - ^ OrthoMaM
Ranwez V, Delsuc F, Ranwez S, Belkhir K, Tilak MK, Douzery EJ (2007). "OrthoMaM: a database of orthologous genomic markers for placental mammal phylogenetics". BMC Evolutionary Biology. 7: 241. doi:10.1186/1471-2148-7-241. PMC 2249597. PMID 18053139.{{cite journal}}: CS1 maint: unflagged free DOI (link) - ^ OrthologID
Chiu JC, Lee EK, Egan MG, Sarkar IN, Coruzzi GM, DeSalle R (March 2006). "OrthologID: automation of genome-scale ortholog identification within a parsimony framework". Bioinformatics. 22 (6): 699–707. doi:10.1093/bioinformatics/btk040. PMID 16410324. - ^ GreenPhylDB
Conte MG, Gaillard S, Lanau N, Rouard M, Périn C (January 2008). "GreenPhylDB: a database for plant comparative genomics". Nucleic Acids Research. 36 (Database issue): D991–8. doi:10.1093/nar/gkm934. PMC 2238940. PMID 17986457. - ^ TreeFam: Tree families database
Ruan J, Li H, Chen Z, et al. (January 2008). "TreeFam: 2008 Update". Nucleic Acids Research. 36 (Database issue): D735–40. doi:10.1093/nar/gkm1005. PMC 2238856. PMID 18056084. - ^ TreeFam: Tree families database
van der Heijden RT, Snel B, van Noort V, Huynen MA (2007). "Orthology prediction at scalable resolution by phylogenetic tree analysis". BMC Bioinformatics. 8: 83. doi:10.1186/1471-2105-8-83. PMC 1838432. PMID 17346331.{{cite journal}}: CS1 maint: unflagged free DOI (link) - ^ Fulton DL, Li YY, Laird MR, Horsman BG, Roche FM, Brinkman FS (2006). "Improving the specificity of high-throughput ortholog prediction". BMC Bioinformatics. 7: 270. doi:10.1186/1471-2105-7-270. PMC 1524997. PMID 16729895.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - ^ Vilella AJ, Severin J, Ureta-Vidal A, Heng L, Durbin R, Birney E (February 2009). "EnsemblCompara GeneTrees: Complete, duplication-aware phylogenetic trees in vertebrates". Genome Research. 19 (2): 327–35. doi:10.1101/gr.073585.107. PMC 2652215. PMID 19029536.
- ^ Sayers EW, Barrett T, Benson DA, et al. (January 2011). "Database resources of the National Center for Biotechnology Information". Nucleic Acids Research. 39 (Database issue): D38–51. doi:10.1093/nar/gkq1172. PMC 3013733. PMID 21097890.
- ^ Zakany, Jozsef; Duboule, Denis (2007-08-01). "The role of Hox genes during vertebrate limb development". Current Opinion in Genetics & Development. 17 (4): 359–366. doi:10.1016/j.gde.2007.05.011. ISSN 0959-437X. PMID 17644373.